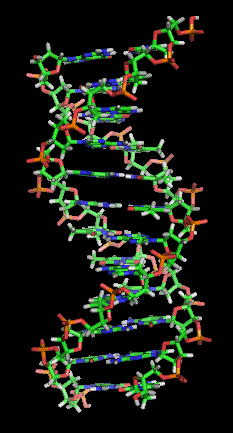
 CRONACA – La rassicurante e lineare rappresentazione del DNA composto da geni che codificano pezzetto per pezzetto tutto ciò di cui siamo costituiti (proteine insomma), alla quale siamo abituati fin dai libri di biologia delle medie è forse destinata a essere rivoluzionata in breve tempo. E diciamocelo c’era da aspettarselo. Se non altro perché almeno dalla pubblicazione dei risultati del progetto Genoma Umano nel 2000 sapppiamo per certo che circa il 98% del nostro DNA apparentemente non ha alcuna funzione (non codifica proteine) e dunque è stato forse frettolosamente chiamato “spazzatura”. Dato però che la Natura è una massaia che non butta via nulla e non spende energie a vuoto, c’era da aspettarsi che questa gran massa di materiale immaginato inerte non potesse essere poi così inutile.
CRONACA – La rassicurante e lineare rappresentazione del DNA composto da geni che codificano pezzetto per pezzetto tutto ciò di cui siamo costituiti (proteine insomma), alla quale siamo abituati fin dai libri di biologia delle medie è forse destinata a essere rivoluzionata in breve tempo. E diciamocelo c’era da aspettarselo. Se non altro perché almeno dalla pubblicazione dei risultati del progetto Genoma Umano nel 2000 sapppiamo per certo che circa il 98% del nostro DNA apparentemente non ha alcuna funzione (non codifica proteine) e dunque è stato forse frettolosamente chiamato “spazzatura”. Dato però che la Natura è una massaia che non butta via nulla e non spende energie a vuoto, c’era da aspettarsi che questa gran massa di materiale immaginato inerte non potesse essere poi così inutile.
Sia chiaro, non è che nessuno si fosse insospettito e in questi anni c’è stato un gran lavoro di ricerca sull’argomento che già aveva iniziato a sgretolare questa concezione. Così ha fatto anche il progetto ENCODE (Encyclopedia of DNA elements) e lo ha fatto in maniera estensiva e sistematica, senza precedenti. Infatti ieri il progetto ha partorito ben 30 articoli scientifici ripartuiti fra le riviste Nature, Science, Genome Biology e Genome Research. Durato cinque anni ha coinvolto 442 ricercatori di 32 istituti sparsi in tutto il mondo (per dare un idea dello sforzo basti sapere che sono stati usati 300 anni di tempo-computer totali per l’analisi dei dati), analizzando 147 tipi di cellule provenienti da diversi tessuti. Risultato: il DNA spzzatura serve a regolare l’azione dei circa 20.000 geni riconosciuti come codificanti del nostro DNA.
La regolazione avviene per esempio sull’accensione e lo spegnimento dei geni codificanti. Più o meno tutte le cellule del nostro corpo contengono infatti l’intera sequenza del DNA che ci caratterizza. Ciononostante, una cellula del fegato, per esempio, e una del cervello esprimono funzioni e proteine diverse. Deve dunque esistere un meccanismo che regola queste differenze e gli scienziati hanno sempre pensato che questo si trovasse all’interno den DNA stesso. Mancava però una visione d’insieme, che ora ENCODE ha fornito.
Secondo i risultati complessivi del progetto circa l’80% del genoma è biochimicamente attivo, in diverse maniere. In alcuni casi le porzioni di DNA servono da target per proteine che influenzano l’attività dei geni, in altri casi codificano RNA che esplica funzioni sue proprie, e altre sono luoghi dove avvengono modifiche chimiche che rendono inattive parti dei cromosomi.
Quello che emerge dagli studi di ENCODE è una rappresentazione molto più complessa del DNA di quella dipinta fino adesso (geni codificanti + junk DNA): oltre ai più di 20.000 geni codificanti conterrebbe unità funzionali di DNA (possiamo chiamare geni anche queste?) che codificano circa 8.800 piccoli RNA, e 9.600 RNA lunghi non codificanti, e 11.224 porzioni di DNA che gli scienziati di ENCODE hanno chiamato pseudogeni, che hanno una qualche funzione biochimica.
Sono stati identificati quattro milioni di “interruttori” che controllano l’attività dei geni, che servono proprio a dare a ciascuna cellula la sua identità a seconda del tessuto in cui si trovano.
Molta attenzione è stata data da ENCODE alla comprensione di certe malattie su base genetica che rappresentano oggi un vero e proprio mistero. Per esempio la malattia di Chron (un’infiammazione del apparato digestivo di origine genetica) che secondo i nuovi risultati dipenderebbe da difetti nel meccanismo di regolazione e non direttamente nei geni codificanti. Molte altre potrebbero essere le malattie che coinvolgono questi meccanismi.
Qui si può trovare una mappa che illustra il ruolo delle diverse porzioni di DNA identificato da ENCODE.