Il DNA sintetico che cresce autonomamente e si auto-assembla
La nuova tecnologia apre la strada a diverse applicazioni, dai DNA-origami ai registratori molecolari.
SCOPERTE – Arrivano novità interessanti dal settore più futuristico della robotica molecolare. Ingegneri del Wyss Institute for Biologically Inspired Engineering presso la Harvard University hanno progettato sequenze di DNA sintetiche in grado di crescere in modo autonomo e di auto-assemblarsi. Le molecole sono in grado di costituire strutture capaci di percepire, amplificare, registrare o valutare logicamente i segnali ambientali. Le sequenze nucleotidiche progettate dal gruppo guidato da Peng Yin potranno costituire la base per una nuova generazione di dispositivi molecolari programmabili. Le strutture sperimentali sono descritte sulle pagine di Nature Chemistry.
Il lavoro è solo l’ultimo prodotto di un filone di ricerca bioingegneristico in forte sviluppo negli ultimi anni, che sta portando a rivalutare il DNA quale materiale “intelligente” e stabile di auto-assemblaggio per costruire nanostrutture e dispositivi molecolari con elevata sensibilità all’ambiente circostante. Capaci per esempio di rilevare infiammazioni nel corpo o tossine nell’ambiente esterno. Tuttavia, finora i ricercatori non potevano fare affidamento su tecnologie in grado di favorire l’accrescimento autonomo di lunghi filamenti singoli di DNA e la loro connessione in modo da poter dare forma a strutture complesse e dispositivi con funzionalità sofisticate.
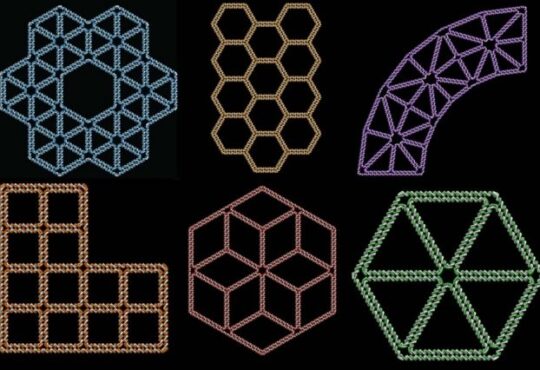
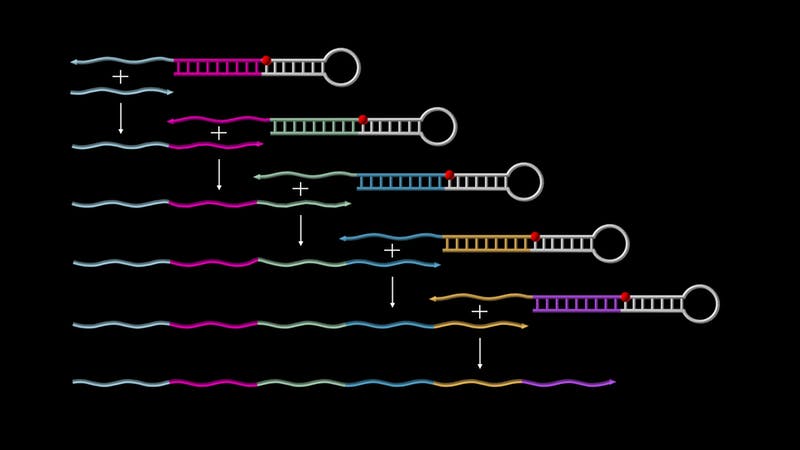
La ricerca di Yin permette di fare fronte a questo problema fornendo soluzioni pratiche per realizzare una nuova generazione di dispositivi molecolari programmabili, costituiti da singole catene nucleotidiche pre-progettate, in grado di crescere in modo autonomo e di formare catene lungo percorsi di montaggio specifici. I ricercatori hanno battezzato la loro nuova tecnologia con il nome di Primer Exchange Reaction (PER) cascades. Le sequenze ottenute sperimentalmente con il metodo PER sono costituite da migliaia di nucleotidi, i mattoncini fondamentali del DNA, contenenti tutte e quattro le sue basi azotate, ossia adenina, timina, citosina, guanina.
Le PER cascades possono essere descritte come una sorta di robot molecolari capaci di eseguire un compito ben determinato a una precisa temperatura. “Stiamo lavorando per implementare le PER cascades per una varietà di applicazioni, tra cui i registratori molecolari, la diagnostica sofisticata e l’imaging dei tessuti”, ha dichiarato in un comunicato Jocelyn Kishi, tra gli autori dello studio. “Ci auguriamo anche che un giorno questi sistemi potranno essere utilizzati in cellule viventi come dispositivi che possono registrare eventi o riprogrammare il comportamento cellulare in modi specifici”.
A parlare della nuova scoperta è anche Donald Ingber, direttore del Wyss Institute. “Questo nuovo avanzamento, che mostra come le molecole di DNA possono essere programmate per auto-assemblarsi in strutture 3D specifiche e svolgere funzioni predefinite, rappresenta un grande passo avanti nel campo della robotica molecolare”.
Il gruppo del Wyss Institute ha sfruttato la nuova tecnologia per progettare una serie di applicazioni concrete molto diverse, tra cui i DNA-origami, grandi nanostrutture di DNA in grado di assemblarsi in modo autonomo, così come approcci di biologia sintetica utili in oncologia. Le sequenze PER potranno essere utili per individuare altre sequenze di DNA e RNA, agendo con un’azione catalitica in grado di tagliare le sequenze in zone bersaglio specifiche, o potranno funzionare come sonde a fluorescenza capaci di amplificare la presenza di uno stimolo ambientale particolare, o anche come registratori molecolari, con la facoltà di indicare l’ordine progressivo in cui determinati segnali molecolari appaiono nel loro ambiente.
Leggi anche: Un “salto nel buio” per curare le infezioni batteriche
Pubblicato con licenza Creative Commons Attribuzione-Non opere derivate 2.5 Italia. ![]()