Una ricerca nata a Trieste ha studiato il meccanismo con cui gli enzimi di restrizione interagiscono con il DNA, per lo sviluppo di biosensori su scala nanometrica.
Una ricerca nata a Trieste ha studiato il meccanismo con cui gli enzimi di restrizione interagiscono con il DNA, per lo sviluppo di biosensori su scala nanometrica.
NOTIZIE – L’immobilizzazione del DNA su supporti solidi, attraverso un processo di auto-assemblaggio e manipolazioni enzimatiche, è un’applicazione importante per lo sviluppo di dispositivi miniaturizzati innovativi per la diagnostica.
Sulla rivista Nature Communications un team interdisciplinare di ricercatori ha recentemente pubblicato i risultati di uno studio che illustra i meccanismi con cui gli enzimi di restrizione interagiscono con il DNA in nanostrutture molecolari. La scoperta può aprire nuovi scenari per la medicina molecolare di frontiera e favorire lo sviluppo di nanotecnologie a basso costo, usando strutture composte da molecole di DNA, utili per esempio per l’analisi di tessuti biologici.
Gli enzimi di restrizione sono uno degli strumenti alla base dell’ingegneria genetica e delle tecniche del DNA ricombinante, tanto importanti da valere un premio Nobel. Nel 1978 infatti, Werner Arber, Daniel Nathans e Hamilton Smith ricevettero il premio Nobel per la Medicina
“for the discovery of restriction enzymes and their application to problems of molecular genetics”.
Questa classe di enzimi è così importante perchè in condizioni fisiologiche riesce a leggere il codice del DNA e a tagliarlo in corrispondenza di specifiche sequenze di nucleotidi. Nei laboratori si sfrutta questa caratteristica per manipolare il DNA e analizzare i geni.
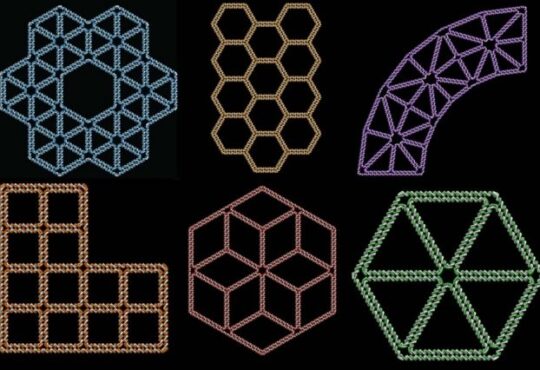
Il gruppo di ricercatori, coordinato da Giacinto Scoles, professore alla Sissa fino al novembre scorso e responsabile, assieme a Loredana Casalis, del laboratorio Senil (Sissa Elettra NanoInnovation Laboratory) di Elettra, ha scoperto come confinare gli enzimi nello spazio bidimensionale, creando strutture di densità variabile di DNA.
In pratica, hanno creato delle vere e proprie “foreste” di DNA, attaccando corte sequenze su una superficie solida, per osservare poi l’azione dell’enzima di restrizione con un microscopio a forza atomica. Hanno scoperto che quando la densità dei paletti di DNA è sufficientemente alta da formare una matrice ordinata, gli enzimi possono accedervi solo dai suoi bordi laterali, mediante diffusione.
Come spiega Matteo Castronovo, in Senil fino al 2008, poi ricercatore presso il Centro di biomedicina molecolare di Trieste, e adesso professore a Temple University (PA, USA), principal investigator della ricerca oggi pubblicata su Nature Communications “Abbiamo individuato che all’interno delle matrici, gli enzimi diffondono bidimensionalmente: cioè gli enzimi attraversano la matrice da un lato all’altro restando intrappolati al suo interno, anche per centinaia di micrometri, attraversando distanze centinaia o decine di migliaia di volte il loro diametro”.
“Con il nostro studio, per la classe di enzimi di restrizione, è come se avessimo scoperto che costruendo un edificio con solo 10.000 frammenti di DNA è possibile isolarlo dall’esterno con delle pareti o un tetto, ma anche munirlo di porte o finestre per comunicare in modo controllato. Si apre così la possibilità di costruire dei veri e propri circuiti (o labirinti) molecolari” spiega ancora Castronovo.
Ricerche di questo richiedono conoscenze non solo di fisica, ma anche di biologia molecolare, medicina, ingegneria chimica ed elettronica, per questo il successo del progetto è il frutto della proficua collaborazione di ricercatori provenienti da ambiti disciplinari molto diversi che lavorano presso la Sissa, la Sincrotrone Trieste, il Centro di Biomedicina Molecolare, l’Ospedale di Cattinara, l’Università di Ancona e quella di Temple negli Stati Uniti.