SAM, così si assembla il DNA. La scoperta di Marco de Vivo, vincitore del premio dell’Acs
Utilizzare i supercomputer per svelare il meccanismo di polimerizzazione del DNA e dell’RNA. Marco De Vivo, ricercatore dell’Iit, ci spiega come ha guidato il suo team alla scoperta del SAM (Self-activated mechanism) pubblicando un articolo sulla rivista Jacs e diventando il primo italiano a vincere il prestigioso premio OpenEye Outstanding Junior Faculty Award della American Chemical Society (Acs)
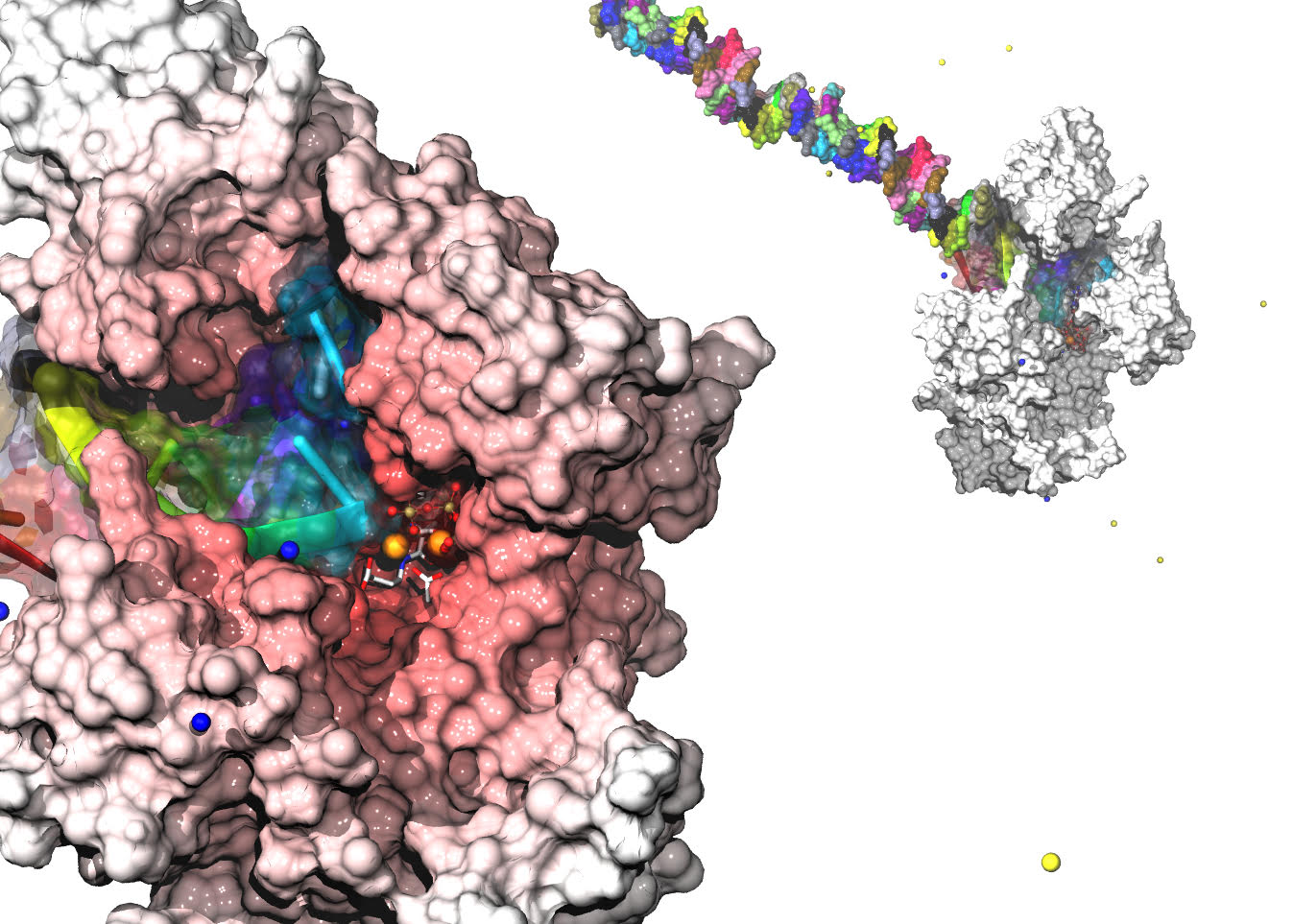
RICERCA – Gli acidi nucleici come RNA e DNA sono soggetti a replicazione. Il codice genetico viene così duplicato e forma lunghe catene a elica nel processo detto polimerizzazione. Ma cosa accade quando i nucleotidi vengono riprodotti e i filamenti iniziano a inanellarsi uno all’altro, mattone dopo mattone? Se fino a oggi gli studi relativi ai meccanismi di duplicazione e replicazione di molecole come DNA e RNA erano “limitati” alla cristallografia, cioè a fotografie delle singole fasi, con la chimica computazionale, che si avvale di potenti supercomputer, i ricercatori sono in grado di vedere anche la dinamica del meccanismo, cioè di osservare con una enorme lente di ingrandimento attimo per attimo cosa accade al filamento.
Proprio grazie all’uso dei supercomputer Marco De Vivo, chimico originario di Rimini che ha lavorato nei più prestigiosi laboratori europei e americani, prima di tornare in Italia all’Istituto Italiano di Tecnologia, Iit, dove dal 2009 è responsabile del gruppo di ricerca Molecular Modeling & Drug Discovery, è riuscito insieme al suo team a svelare il Self-activated mechanism, SAM, cioè il meccanismo di polimerizzazione, cioè di allungamento dell’acido nucleico, sia esso DNA, RNA o XNA, il DNA sintetico. Il risultato di De Vivo è stato pubblicato in un articolo sulla rivista scientifica Jacs ed è valso al ricercatore il prestigioso premio OpenEye Outstanding Junior Faculty Award dell’American Chemical Society, facendo di lui il primo italiano a vincere questo riconoscimento per il proprio lavoro.
Ma che cos’è la SAM e perché è così importante?
Nel loro ciclo vitale gli acidi nucleici come RNA e DNA devono essere allungati e polimerizzati. Immaginate di voler costruire una sequenza di mattoncini, i nucleotidi. Esistono degli enzimi che fanno questo “mestiere”, cioè quello di attaccare il nuovo nucleotide al successivo, facendo così crescere il filamento di DNA o RNA. Il self-activated mechanism, lo dice il nome, è un meccanismo di polimerizzazione, cioè un allungamento dell’acido nucleico che funziona sia nel processo di replicazione delle cellule sane che per quelle malate. Tutto quello che sappiamo oggi su questo meccanismo viene dalla cristallografia, una sorta di macchina fotografica che scatta delle immagini del sistema in diversi attimi. Poi, con gli esperimenti, descriviamo gli aspetti cinetici, ma in maniera indiretta ed empirica.
Con l’uso dei supercomputer, invece, c’è una vera e propria rivoluzione. E’ come disporre di una enorme lente di ingrandimento che ci permette di vedere attimo dopo attimo cosa accade e come gli enzimi catalizzano, cioè velocizzano, la reazione. Il meccanismo che abbiamo scoperto spiega molti dati sperimentali che prima non potevamo connettere in modo semplice, perché avevamo solo dati statici del ciclo catalitico. Ora invece siamo stati in grado di osservare la reazione dall’inizio alla fine, cioè fino a quando il singolo mattoncino viene aggiunto al filamento e la macchina è pronta per un altro ciclo, che dunque si auto-sostiene. Proprio da questo suo auto-sostenersi viene il nome di self-activated mechanism, SAM.
Uno studio innovativo che per la prima volta mette insieme due campi vicini ma diversi: quello della chimica della reazione con l’aspetto fisico, cioè la sua dinamica.
La comprensione di questo meccanismo apre la strada ad una nuova serie di studi che puntano ad unire sia gli aspetti chimici, inerenti dunque alla reazione, che gli aspetti fisici, necessari affinché il mattoncino si leghi nella posizione giusta e il filamento scorra, in modo che ci sia un inanellamento di mattoncini in un loop che permette di avere un ciclo perpetuo come avviene con i sistemi di polimerizzazione di RNA e DNA.
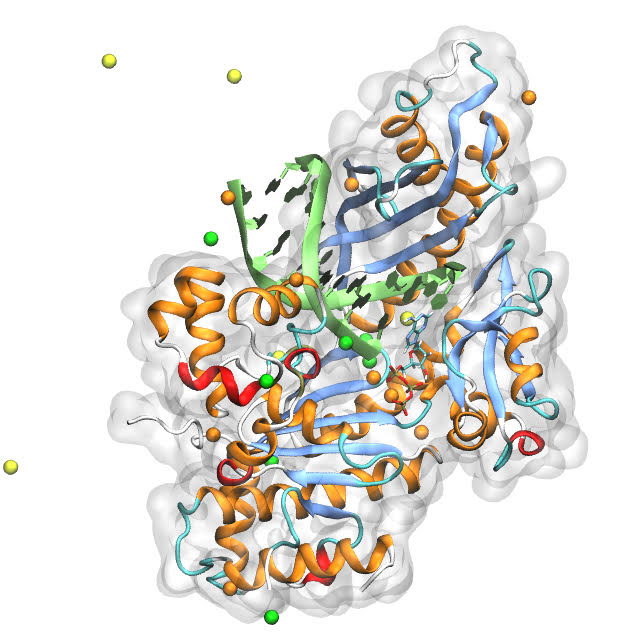
Tra le implicazioni di questa importante scoperta c’è sicuramente un passo avanti nelle terapie personalizzate.
Il concetto di base della medicina personalizzata è che ognuno di noi ha un diverso corredo genetico che lo porta a rispondere alla stessa malattia, ad esempio in caso di tumore, in modo assai diverso al farmaco che gli viene somministrato. Ad esempio, alcuni pazienti possono avere una mutazione che rende i loro enzimi più reattivi alle cure. Altri invece hanno una mutazione che, al contrario, rende la risposta al farmaco nulla. Tracciando il profilo genetico del paziente dunque è possibile capire qual è il farmaco con più alta risposta e, in futuro, riuscire a fornire una terapia che sia personalizzata e più efficace, mentre oggi i farmaci vengono somministrati in modo indiscriminato, senza cioè tenere conto delle eventuali mutazioni genetiche. Capire meccanismi come il SAM quindi rappresenta un passo importante nella comprensione della replicazione del DNA e quindi offre una possibilità concreta di agire sul meccanismo stesso.
Conoscere nel dettaglio questo meccanismo, che è estremamente elegante e che permette di spiegare dati che fino ad oggi non erano stati razionalizzati, permetterà di capire anche come modularlo o bloccarlo, un domani, nelle cellule tumorali, in modo da fermarne la replicazione. Conosciamo già RNA e DNA, ma è molto importante cercare di capire nel dettaglio come le differenze tra i nostri profili genetici sono in grado di gestire la generazione delle proteine e quindi tutti i possibili problemi che ne seguono.
Non solo terapie personalizzate, la comprensione della SAM apre anche allo studio dell’Xna, il DNA sintetico in cui le basi azotate come adenina, guanina, timina e citosina, vengono sostituite con altre basi sintetiche.
Si tratta di forme sintetiche di DNA che vengono costruite su delle basi sintetiche, che non sono le classiche basi azotate, e che permettono di costruire delle eliche come nel caso del DNA. L’XNA rappresenta a oggi una nuova frontiera della genetica ed è un campo del tutto agli albori, dalle applicazioni infinite e, a oggi, difficili da immaginare. Ci offre uno strumento unico per emulare la natura e creare un sistema che poi esprima proteine in maniera controllata. Comprendere il funzionamento dei meccanismi che regolano la replicazione degli acidi nucleici è necessario per poter in futuro modulare o bloccare il processo endogeno che scopriamo nocivo.
Il risultato raggiunto con il suo team mette in evidenza l’importanza della chimica computazionale, che è in grado di abbattere i tempi e i costi della progettazione di molecole e farmaci. In media infatti dalla progettazione della molecola all’arrivo del farmaco agli utenti ci vogliono 12 anni di lavoro e investimenti da 1,5 milioni di euro per arrivare ad un risultato.
La chimica computazionale ha totalmente rivoluzionato la “drug discovery“, cioè la scoperta di nuovi farmaci. Fino a qualche anno fa si andava avanti per tentativi: il ricercatore si basava sulla propria esperienza e tentativo dopo tentativo si trovavano le molecole. Oggi invece si affronta uno step precedente di progettazione al computer, come si fa con le case. Prima si verifica che le fondamenta tengano, poi che il tetto non sia soggetto a crolli e una volta che il progetto è concluso si procede alla costruzione.
Proprio come per la progettazione delle case, le molecole vengono prima progettate al computer. In questo modo possiamo focalizzare i test e scegliere il set di molecole più promettenti che saranno poi testate in laboratorio, escludendo invece quelle che nelle simulazioni hanno già mostrato che è altamente improbabile che portino ad un risultato. In questo modo si va ad agire su due aspetti della sperimentazione. In primo luogo si abbassano notevolmente i costi, dato che si investe solo sulle molecole che è più probabile che diano il risultato sperato, e poi si velocizzano i tempi grazie alla razionalizzazione degli esperimenti, con un conseguente aumento del grado di successo.
Si parla molto di cervelli in fuga, ma lei è tornato in Italia dopo aver lavorato per oltre cinque anni nelle migliori università e laboratori europei e degli Stati Uniti.
Sono orgoglioso e grato del premio ricevuto dall’Acs e per questo devo ringraziare il mio team, la mia famiglia e l’Iit che in Italia rappresenta un istituto di eccellenza. Parlare di cervello in fuga, però, non è corretto. Per un ricercatore è assolutamente indispensabile fare esperienze all’estero, perché è fuori che si costruiscono le conoscenze e i network di ricercatori con cui confrontarsi quando si svolge il mestiere del ricercatore. Per fare questo lavoro dobbiamo confrontarci con la realtà non solo italiana, ma mondiale. Quello che però conta è riuscire a rendere l’Italia attrattiva per un ritorno, non solo dei giovani ricercatori italiana, ma anche degli stranieri, perché è solo con l’investimento di risorse, umane ed economiche, che possiamo raggiungere grandi risultati nella ricerca.
Leggi anche: Il gene riluttante: diamo troppe responsabiltà al DNA?
Leggi anche: Nuove frontiere dell’editing genetico





