La rivoluzione delle strutture proteiche 3D
Che cos'è la microscopia crioelettronica e a cosa serve? Ne parliamo con Giuseppe Cannone.
RICERCANDO ALL’ESTERO – La scorsa settimana è stato assegnato il premio Nobel 2017 per la chimica a Jacques Dubochet, Joachim Frank e Richard Henderson per il loro lavoro sulla microscopia crioelettronica. Ne abbiamo parlato in questo articolo di approfondimento.
Henderson ha condotto le sue ricerche al Laboratorio di biologia molecolare del MRC di Cambridge (Regno Unito); Giuseppe Cannone è uno dei responsabili del microscopio crioelettronico del MRC, fa parte del Research Support Office e collabora con diversi gruppi di ricerca per definire la struttura di complessi di proteine.
Quali sono i vantaggi della microscopia crioelettronica?
Innanzitutto il fatto che la molecola di interesse non deve essere cristallizzata. In passato il principale metodo per definire la struttura di complessi proteici era la cristallografia; tuttavia riuscire a ottenere il cristallo di una proteina non è molto semplice perché bisogna individuare le condizioni sperimentali più adatte e possono volerci addirittura decenni.
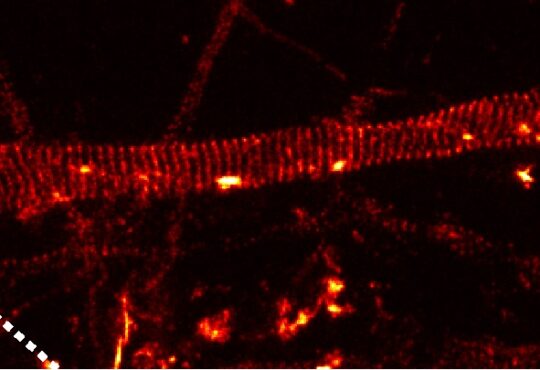
Grazie alla microscopia crioelettronica (crio-EM) tutto il processo è più veloce: dopo essere stata estratta e purificata, la proteina rimane in un ambiente molto simile a quello fisiologico e non subisce alterazioni né di struttura né di orientamento nello spazio. In questo senso il ghiaccio vitreo è un ottimo materiale, contiene tutti gli elementi che rendono la proteina felice di stare in soluzione ed è trasparente, cosa che aumenta il contrasto dell’oggetto da studiare. Al microscopio le proteine non hanno un grande contrasto e per migliorarlo si possono usare diversi materiali, ma molti di quelli in uso modificano la disposizione della proteina nello spazio e ne alterano la struttura fisiologica.
Come si produce il ghiaccio vitreo?
Le proteine in soluzione vengono applicate a un supporto, chiamato griglia, che viene immediatamente congelato in etano liquido a una temperatura di -180 °C. Il processo di congelamento è così rapido che l’acqua forma direttamente un solido amorfo trasparente senza passare attraverso gli stadi cristallini: i cristalli d’acqua, infatti, hanno un contrasto maggiore e interferirebbero con l’acquisizione dell’immagine. Una volta vetrificato, il campione viene caricato sul microscopio a trasmissione elettronica, precedentemente raffreddato a circa -190 °C; man mano che gli elettroni attraversano le proteine si creano delle proiezioni che vengono registrate da una fotocamera digitale. Si ottengono decine di migliaia di immagini per lo stesso oggetto ma tutte con orientamento diverso, come se le proteine facessero delle capriole nel ghiaccio.
Il numero di fotografie, chiamate micrografie, dipende da quanto tempo si ha a disposizione al microscopio: in 24 ore si possono raccogliere circa 1000 micrografie con un ingrandimento tra 50 mila e 100 mila volte. Ogni micrografia contiene 50-100 particelle, cioè complessi proteici. Grazie a specifici algoritmi, le particelle vengono prima isolate e poi allineate una a una per selezionare quelle con la stessa disposizione nello spazio; le immagini “simili” vengono sommate a formare le cosiddette class averages, indispensabili per aumentare il contrasto della proteina che si trova in quella determinata posizione. Le circa 300-400 particelle ottenute hanno un contrasto esagerato e possono venir processate da ulteriori algoritmi che calcolano le relazioni tra le diverse proiezioni.
Sulla base di centinaia di immagini bidimensionali, quindi, si può ricostruire un volume tridimensionale che permetterà di definire la struttura del complesso proteico di interesse.
Questa tecnica può essere usata anche per singole proteine?
Sì, grazie all’avanzamento tecnologico dei microscopi e ai miglioramenti delle fotocamere digitali è possibile oggi ottenere una risoluzione alta anche per singole proteine.
Fino a cinque anni fa, per esempio, era impossibile definire la struttura dell’emoglobina tramite microscopia elettronica mentre le attuali tecnologie ci hanno permesso finalmente di vederla nel ghiaccio.
Il problema degli oggetti piccoli, infatti, è riuscire a visualizzarli perché si confondono con il background e il software fornisce falsi positivi dato che non riesce a elaborare in maniera corretta i calcoli statistici. In generale, più l’oggetto è grande, maggiore è il contrasto e migliore è l’elaborazione digitale delle immagini.
La crioEM rappresenta una grande rivoluzione e un gigantesco passo avanti per tutte quelle proteine che non si riescono a cristallizzare: riuscire a visualizzarle permetterà di studiarne la funzione, sia in condizioni fisiologiche sia in caso di mutazioni o malattie.
Che tipo di proteine stai studiando grazie alla microscopia crioelettronica?
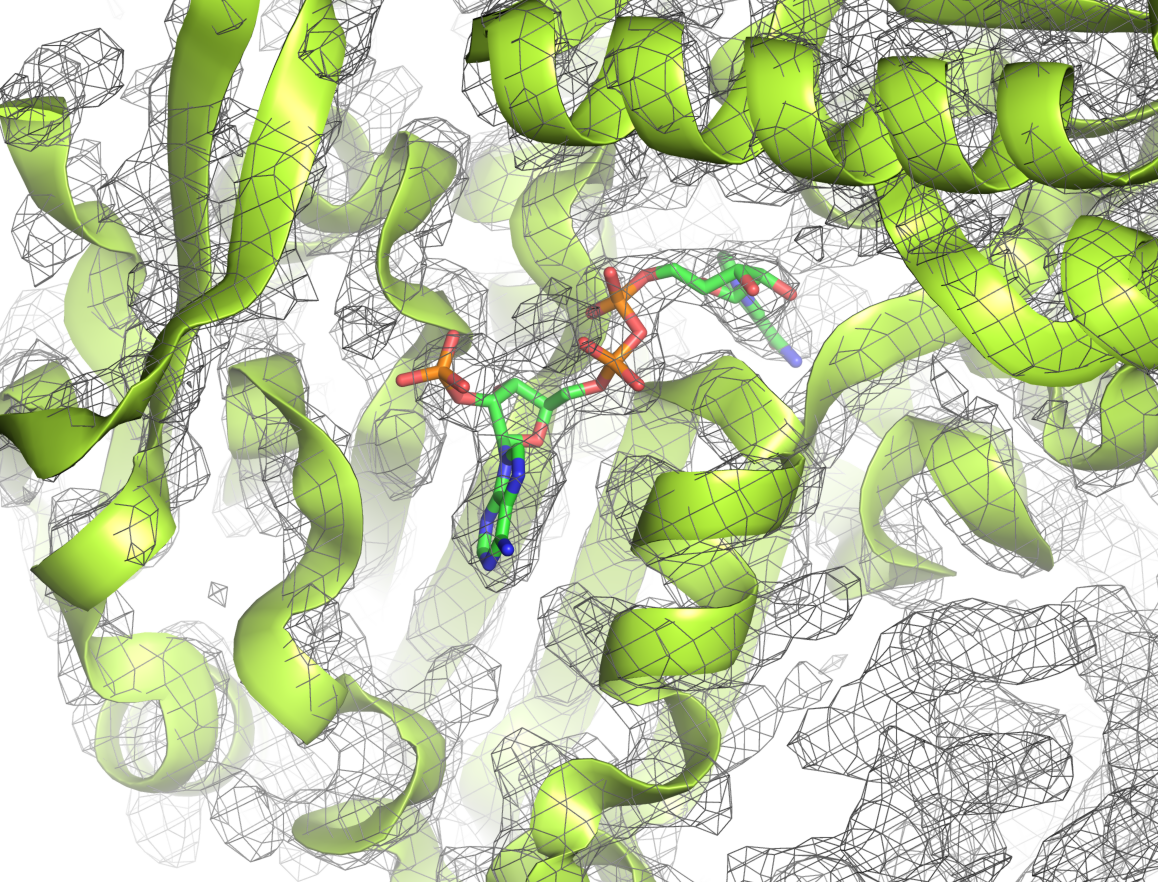
Sto collaborando con diversi gruppi, di recente ho iniziato un progetto per definire la struttura molecolare di un complesso coinvolto nella replicazione del DNA eucariotico, in particolare nello svolgimento della doppia elica. Il complesso è formato da diverse proteine: MCM (Mini Chromosome Maintenance) è una specie di ciambella che ruota attorno al DNA e lo apre in due singole eliche; per funzionare, MCM ha bisogno di altri partner tra cui Cdc45, GINS, Mrc1, Ctf4 e Tof1.
Il nostro obiettivo è capire come sono organizzate tutte queste molecole nello spazio, quali sono le superfici di interazione tra i vari componenti, quali sono gli amminoacidi più coinvolti.
Saperlo è interessante sia come ricerca di base sia per future applicazioni nel campo delle terapie anticancro: molti dei farmaci chemioterapici agiscono proprio a livello della replicazione del DNA, dato che le cellule tumorali si replicano molto velocemente e in maniera incontrollata. Conoscere i dettagli sulle interazioni che legano le proteine coinvolte nella replicazione permetterebbe, per esempio, di sviluppare dei farmaci che distruggono tali interazioni.
Un altro progetto a cui mi sto dedicando riguarda la struttura di un enzima coinvolto nella degradazione di composti aromatici. Paaz è una proteina bellissima che rompe l’anello benzenico producendo acqua ed energia; dal punto di vista biochimico, questa rottura richiede un’energia molto alta ma Paaz lo fa in maniera molto elegante, attraverso due reazioni e in pratica recuperando energia. Dal punto di vista biotecnologico, riuscire a implementare una reazione del genere in bioreattori vorrebbe dire degradare composti tossici per l’uomo a impatto ambientale zero.
Quali sono le prospettive future del tuo lavoro?
L’ultimo progetto a cui sto lavorando è top secret. A parte gli scherzi, posso solo dire che stiamo cercando di definire la struttura di un recettore accoppiato alle proteine G.
 Nome: Giuseppe Cannone
Nome: Giuseppe Cannone
Età: 33 anni
Nato a: Cerignola (FG)
Vivo a: Cambridge (Regno Unito)
Dottorato: biologia molecolare e strutturale (Edimburgo, Regno Unito)
Ricerca: Definire i dettagli molecolari delle proteine attraverso la criomicroscopia elettronica
Istituto: Laboratory of molecular biology, Medical Research Council (Cambridge, UK)
Interessi: fotografia, sport, leggere
Di Cambridge mi piace: la campagna
Di Cambridge non mi piace: il clima, non c’è il sole
Pensiero: La scienza è come il sesso: non c’è dubbio che facendola si ottengano dei risultati pratici, ma non è per quello che la si fa. (Richard Feynman)
Leggi anche: Nobel per la Chimica 2017 agli inventori della microscopia crioelettronica
Pubblicato con licenza Creative Commons Attribuzione-Non opere derivate 2.5 Italia. ![]()